Características y funcionamiento de los Sistemas de Dos Componentes de organismos procariotas y eucariotas
Adrián F. Álvarez y Dimitris Georgellis
Departamento de Genética Molecular, Instituto de Fisiología Celular, Universidad Nacional Autónoma de México
Ciudad de México. México.
Resumen
Los Sistemas de Dos Componentes (SDCs) son circuitos de señalización que permiten a los organismos tanto procariotas como eucariotas detectar estímulos ambientales y responder a estos de manera adaptativa. Estos sistemas de transducción de señales, que se basan en la transferencia de grupos fosforilo entre residuos de histidina y aspartato de una proteína histidín- quinasa y una reguladora de respuesta, se caracterizan por tener una alta especificidad que garantiza la repuesta adecuada a cada nueva condición. Aunque muchos SDCs co-existen en cada célula bacteriana, diferentes mecanismos evitan fosforilaciones cruzadas que pondrían en riesgo dicha especificidad. Sin embargo, existen conexiones funcionales entre SDCs, que se conocen como regulación cruzada, que favorecen la modulación de una respuesta celular coordinada ante la presencia de múltiples estímulos. Curiosamente, los SDCs de células eucariotas, a pesar de que conservan algunas propiedades de los sistemas bacterianos, adquirieron arquitecturas y en muchos casos modos de acción diferentes a estos. En esta revisión, además de exponer las características estructurales y funcionales más importantes de los SDCs bacterianos, realizamos una comparación de las arquitecturas de los SDCs en células procariotas y eucariotas, discutiendo brevemente sobre las consecuencias de las diferencias existentes en la especificidad de dichos sistemas.
Palabras clave: Sistemas de dos componentes, fosforilación, especificidad, señalización.
Characteristics and operation of Two Component Systems of prokaryotic and eukaryotic organisms
Summary
Two-component systems (TCSs) are signaling circuits that allow both prokaryotic and eukaryotic organisms to detect environmental cues and respond to them in an adaptive way. These signal transduction systems, based on phosphoryl-group transfers between histidine and aspartate- containing sensor kinase and response regulator proteins, are characterized to be highly specific in order to assure an appropriate response to each particular condition. Although many TCSs co-exist in a bacterial cell, different mechanisms prevent cross phosphorylation events that could compromise such specificity. However, there are functional connections between different TCSs, called cross regulation, which allows cells to modulate a coordinated response to multiple signals. Curiously, TCSs of eukaryotic cells, although conserve some bacterial TCSs features, acquired new architectures and, sometimes, novel modes of action. In this review, in addition to describe the most important structural and functional characteristics of bacterial TCSs, we compare the TCS architecture of prokaryotes and eukaryotes, and discuss briefly the consequences of the differences on signaling specificity.
Keywords: Two component systems, phosphorylation, specificity, signaling.
Introducción
Todos los organismos tienen la capacidad y la necesidad de comunicarse con el medio que los rodea. Esto es importante principalmente para los microorganismos, cuyo hábitat suele estar sujeto a cambios de manera constante y repentina. En particular las bacterias han desarrollado sistemas modulares que les permiten detectar cambios en las condiciones de su entorno y responder de manera coordinada para adaptarse al nuevo escenario. Cada uno de estos sistemas está conformado a menudo por dos proteínas, por lo que se los conocen como sistemas de dos componentes (SDCs) [1, 2]. Un SDC típico está constituido por una histidín-quinasa (HK) y una proteína reguladora de respuesta (RR), quienes en conjunto le permiten a la célula detectar señales ambientales y responder de manera adaptativa mediante cambios en la expresión génica. Los SDCs constituyen los principales sistemas de transducción de señales en bacterias, y están presentes en casi la totalidad de las mismas [3–5]. Observando y analizando la vasta información genómica disponible, se puede encontrar una correlación entre el número de SDCs existente en una célula bacteriana y la susceptibilidad a cambios en las condiciones del ambiente que este microorganismo habita. Por ejemplo, Pseudomonas aeruginosa, que tiene la capacidad de vivir en múltiples ambientes incluyendo suelo y agua, cuenta con alrededor de 60 sistemas de transducción de señales de dos componentes [4, 6], mientras que Escherichia coli, cuyo hábitat se circunscribe al interior del intestino de mamíferos, tiene la mitad de estos sistemas [4, 7]. Siguiendo esta tendencia, el número de SDCs por célula bacteriana oscila entre 0, como es el caso de algunas especies del género Mycoplasma, que son parásitos intracelulares obligados, hasta los casi 200 que poseen células bacterias del género Streptomyces, que habitan en suelo.
Los SDCs no son exclusivos de las bacterias, ya que también se encuentran presentes en la mitad de los genomas secuenciados de Arqueas [8]. En cuanto a células eucariotas, a pesar de que los SDCs están ausentes en animales y protistas, estos pueden encontrarse en levaduras, hongos, amebas sociales y plantas [9–14]. Curiosamente, los SDCs, típicamente procariotas, han sido adoptados y adaptados por organismos eucariotas para realizar funciones de señalización, pero con notables cambios en arquitectura, organización y funcionamiento con respecto a los sistemas bacterianos [12, 15].
El éxito evolutivo de los SDCs bacterianos se debe a características estructurales y funcionales que iremos describiendo a lo largo de los siguientes párrafos. Algunas de estas características fueron aprovechadas por células eucariotas, mientras que otras han sido notablemente modificadas al ser integradas en redes de señalización propias de estas células. Al respecto, discutiremos algunos aspectos sobresalientes.
Funcionamiento de un SDCs típico
Un sistema canónico de dos componentes está constituido por dos proteínas: una HK y una RR. La HK, que normalmente se encuentra anclada a la membrana citoplasmática, contiene un dominio sensor que es el encargado de detectar la presencia de la/s señal/es que activa/n el sistema, y un dominio transmisor (DT), que consta de un motivo de unión a ATP y de un residuo de histidina (His) que es susceptible de ser fosforilado [2, 16] (Fig. 1). Es generalmente aceptado que este DT actúa también como un dominio de dimerización, ya que todas las HK actúan como homodímeros [17– 20]. Por su parte, una RR tiene a su vez dos dominios: un dominio receptor (DR) que contiene un residuo de aspartato (Asp) conservado, y un dominio efector, que generalmente es un motivo de unión a DNA (Fig. 1). Como veremos a continuación, cada paso involucrado en la señalización a través de un SDC, tiene un alto grado de especificidad. Así, el dominio sensor de una HK es capaz de unir de manera específica a uno o unos pocos ligandos, que constituyen la señal que activa al sistema [21]. De manera que si en una condición determinada de crecimiento está presente la señal que activa a un SDC dado, se habla de que esas son condiciones estimulantes con respecto a ese sistema en particular. Cuando esto ocurre, el domino sensor de la HK detecta la señal, lo que provoca que esta proteína se active como quinasa, auto-fosforilándose en el residuo de His conservado [22]. Posteriormente, el grupo fosforilo (~P) es transferido específicamente al residuo de Asp del dominio receptor de la RR cognada (Fig. 1). Esta proteína, en su estado fosforilado, se une a regiones promotoras de determinados genes mediante el dominio de unión a DNA, activando o reprimiendo la transcripción de los mismos [22]. Como resultado de esta modificación en la expresión génica, las células modifican ciertas características que le permiten adaptarse a las nuevas condiciones ambientales.
Eventualmente, las condiciones del medio volverán a cambiar, y la señal activadora del sistema en cuestión estará ausente. Bajo estas circunstancias, la HK dejará de estar activa como quinasa, y la proteína RR fosforilada (RR-P) perderá el grupo ~P, inactivándose como regulador transcripcional. Frecuentemente, la desfosforilación de la RR está catalizada por la misma HK cognada que, en ausencia de la señal, adquiere una actividad fosfatasa específica de su RR-P [22, 23] (Fig. 1). Como se detalla más adelante, esta característica de las HKs ayuda a mantener la especificidad en la transducción de una señal.
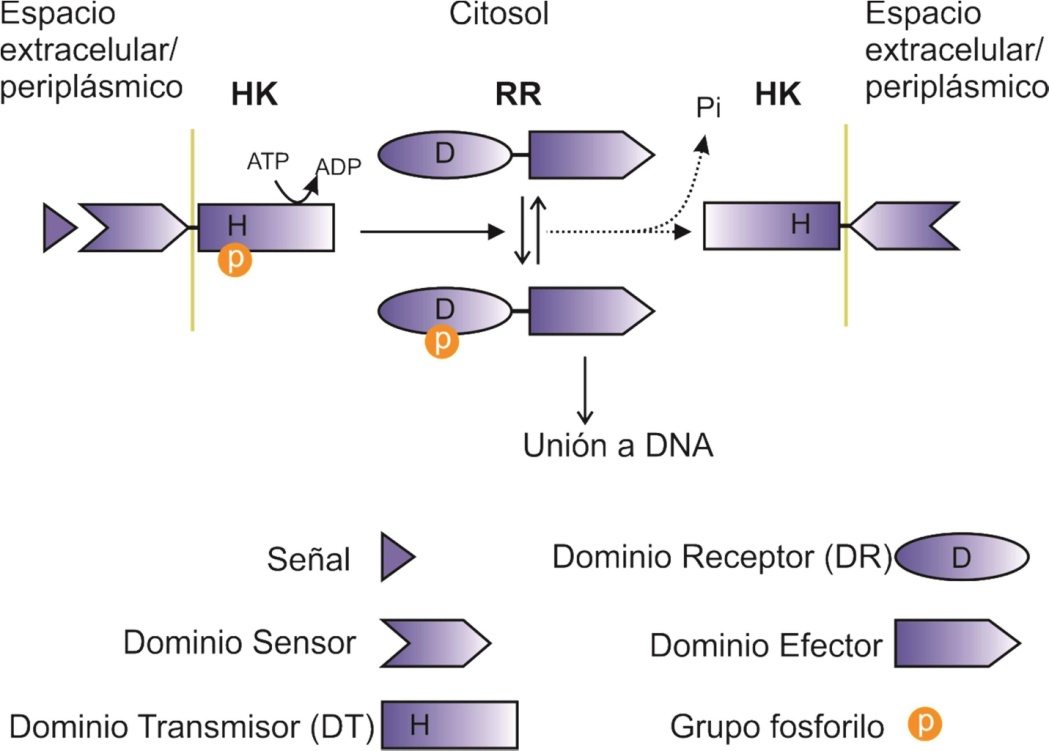
Figura 1: Sistema de dos componentes bacteriano canónico. En presencia de una señal específica (izquierda), una HK anclada a la membrana plasmática cataliza la fosforilación del RR cognado, activándolo como factor transcripcional, mientras que en ausencia de la señal, (derecha) la misma HK cataliza la desfosforilación del RR-P, inactivándolo y silenciando el sistema.
Los SDCs están compuestos por módulos funcionales
En la descripción de un SDC canónico, mencionamos a los dominios transmisor (DT) y receptor (DR), esenciales para la transducción de una señal molecular mediante reacciones de fosforilación/desfosforilación. Algunos SDCs bacterianos, y la mayoría de los que se encuentran en células eucariotas, cuentan con módulos adicionales que participan en la transducción de la señal. Estos son un dominio receptor adicional y un domino de fosfotransferencia (HPt), este último con un residuo de His conservado pero, a diferencia del DT, sin un motivo de unión a ATP (Fig. 2). En bacterias, estos módulos adicionales se encuentran frecuentemente formando parte de una histidín-quinasa híbrida (HHK), también llamada tripartita ya que contiene, además del dominio sensor, tres dominios catalíticos que son el DT, el DR y el HPt. Cuando la señal está presente y es detectada por el dominio sensor de una HHK, el dominio transmisor se autofosforila en el residuo de His conservado, y posteriormente el ~P es transferido secuencialmente al Asp del DR y a la His del HPt de la misma proteína, (Fig. 2), para finalmente fosforilar al Asp del DR de la proteína RR [24–28]. A este proceso se lo conoce como fosforelevo. De la misma manera que ocurre con las HK canónicas, cuando las condiciones cambian nuevamente y el estímulo está ausente, la HHK se inactiva como quinasa y se activa como fosfatasa especifica de su RR cognada. En este caso, la desfosforilación ocurre mediante un fosforelevo inverso, que involucra el pasaje del ~P del DR de la RR a la His del HPt, luego al residuo de Asp del DR de la HHK y finalmente la liberación de fosfato inorgánico (Fig. 2) [29–31]. Cabe destacar, que las transferencias de ~P siempre siguen la sucesión de His a Asp y de Asp a His, y esto obedece a cuestiones energéticas que impide la transferencia de un grupo fosforilo entre dos His o entre dos Asp [22].
Curiosamente, dependiendo del sistema en cuestión, los diferentes dominios de una HHK pueden formar parte de la misma proteína o pueden estar naturalmente distribuidos en péptidos separados, sin perder la especificidad en la transferencia del ~P. Esto permite encontrar sistemas en donde hay HHK tripartitas (con los tres dominios catalíticos que son DT, DR y HPt), otros en donde hay una HHK bipartita (conteniendo DT y RD, con el HPt formando parte de otra proteína), y finalmente existen sistemas en donde los tres dominios se encuentran en proteínas separadas. Los diferentes dominios catalíticos que componen un SDC han evolucionado de manera que cada uno puede operar de manera autónoma, interactuando con los demás módulos específicos aunque no exista un enlace covalente manteniéndolos en cercanía [26, 30, 32]. Esto supone un incremento en la versatilidad de estos sistemas de señalización, lo que podría explicar su amplia distribución no sólo entre las bacterias sino también entre organismos eucariotas.
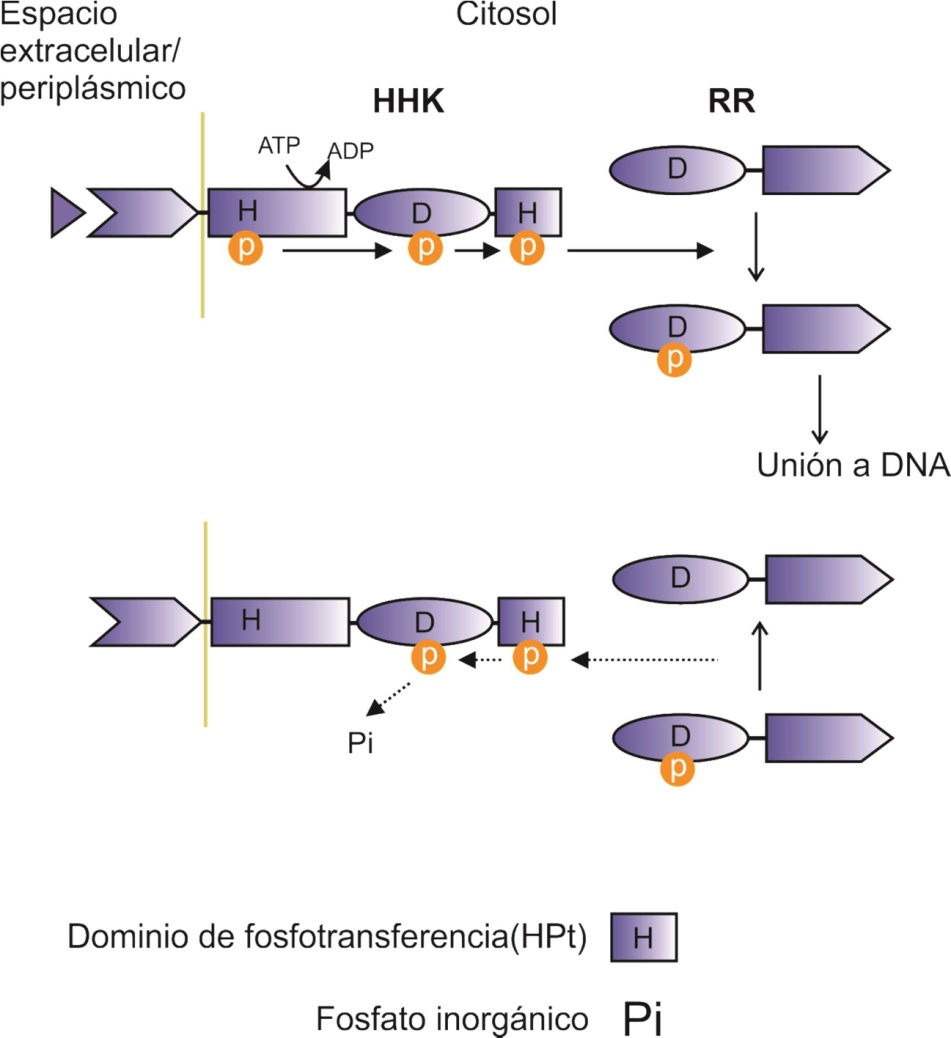
Figura 2: Histidin-quinasas híbridas y fosforelevo. HHKs catalizan la fosforilación del RR cognado mediante un fosforelevo que se caracteriza por la transferencia del grupo fosforilo entre residuos de His y Asp (arriba). En ausencia de la señal, la HHK cataliza la desfosforilación del RR- P mediante un fosforelevo inverso (abajo). Los módulos de una HHK (DT, DR y HPt) pueden estar en la misma proteína o formar parte de proteínas independientes.
Diversidad de estímulos, de dominios sensores y de respuestas
Hasta este momento hemos revisado las características comunes más importantes de los SDCs bacterianos. Sin embargo, la diversidad de estímulos con los que puede y debe contender una célula bacteriana es innumerable. Además, los distintos sistemas han evolucionado a la par de la ocupación de nuevos hábitats por parte de los microorganismos, y esto redunda en una gran variedad de mecanismos de detección de señales y de respuestas celulares. Por consiguiente, el estudio de cada nuevo SDC presenta nuevos retos. Debido a su variabilidad en estructura y localización, los dominios sensores de las HKs se encuentran mucho menos caracterizados que los dominios catalíticos [33, 34]. Si bien una HK típica posee un dominio sensor extracelular o periplásmico, muchas HKs detectan la señal en la región citosólica de la proteína o en los cruces transmembranales.
Un dominio que aparece formando parte de numerosas HKs es un motivo estructural denominado PAS (Per/ARNT/Sim) [35–37], que contiene una típica conformación de hélices a que flanquean cinco láminas þ-plegadas antiparalelas. Sin embargo, sólo en algunos casos puntuales se ha demostrado que este motivo estructural es un dominio sensor funcional, como ocurre en las quinasas PhoQ, DcuS y CitA, que detectan iones divalentes, C4-dicarboxilatos y citrato, respectivamente [38–42]. En estas HKs, la detección de la señal ocurre por interacción directa del ligando (señal) con el dominio estructural PAS que se encuentra en la región extracelular/periplásmica de la proteína. En otros casos, el motivo PAS se asocia con moléculas que actúan como grupo prostético y éste es el que percibe el estímulo. Uno de estos casos es el sistema FixL/FixJ de Sinorhizobium meliloti, una bacteria que establece una relación simbiótica con plantas al formar nódulos en sus raíces y permitir la fijación de nitrógeno. La histidín-quinasa FixL constituye un sensor de oxígeno, ya que contiene un motivo PAS localizado en la región citosólica de la proteína, que une a un grupo hemo-ferroso, que es capaz de detectar la presencia de oxígeno, lo que resulta en la inactivación de FixL como quinasa [43]. A pesar de detectar la señal en la región citosólica de la proteína, FixL de S. meliloti es una proteína integral de la membrana. Sin embargo, las HKs FixL homólogas de Bradyrhizobium japonicum y Rhizobium etli, especies filogenéticamente cercanas a S. meliloti, son proteínas solubles. Existen otras HKs que actúan como detectores de la disponibilidad de oxígeno que cuentan con un mecanismo diferente. Por ejemplo la HK ArcB de E. coli, es parte de un SDC que regula la expresión de más de 300 genes en función de la disponibilidad de oxígeno o del estado redox del medio [44, 45]. ArcB contiene un motivo PAS que, sin embargo, no se une directamente a ningún grupo prostético ni a un ligando específico [46, 47]. En cambio, contiene dos residuos de cisteína que son oxidados, formando puentes disulfuro, en presencia de oxígeno [48]. No obstante, no es el oxígeno la señal del sistema sino las quinonas de la membrana que según su estado de óxido-reducción son capaces de oxidar o reducir las cisteínas de la HK ArcB, inactivándolo o activándolo como quinasa, respectivamente [49, 50].
Si bien el motivo PAS constituye uno de los más abundantes dominios sensores en HKs, se ha reportado que existen al menos 14 dominios sensores diferentes [51]. Incluso hay HKs que no contienen ningún dominio sensor evidente. Por ejemplo la HK DesK, que forma parte del sistema DesK/DesR de Bacillus subtilis, es un sensor de temperatura que cuenta con 5 cruces transmembranales y ningún dominio sensor predicho mediante secuencia o estructura. El cambio en el estado de fluidez de la membrana provocado por cambios en la temperatura es detectado por los cruces transmembranales de DesK, regulando su actividad quinasa/fosfatasa [52]. En este caso, la señal (cambio de temperatura) no es un ligando y es detectada en la región transmembranal de la proteína. Como puede observarse, la identificación del dominio sensor de una HK frecuentemente no es una labor fácil. Por otra parte, la determinación de la naturaleza de la señal de un SDC es muchas veces un proceso complejo, siendo ésta desconocida en la mayoría de los sistemas bacterianos estudiados.
Así como el proceso de detección de la señal de cada HK es único, también la respuesta celular provocada por la activación de un SDC es exclusiva de cada sistema. En la gran mayoría de los casos, la respuesta involucra la regulación de la expresión génica [53], existiendo sistemas que regulan la expresión de unos pocos genes y otros que regulan la expresión de cientos de operones. Además, el domino efector de una RR puede no ser un motivo de unión a DNA, sino que puede estar constituido por una región con actividad enzimática o un dominio de interacción con otras proteínas [53]. En estos casos, la respuesta celular a un estímulo específico no es la regulación transcripcional directa.
Especificidad de los SDCs
Dependiendo de la especie, cada célula bacteriana contiene al mismo tiempo varias decenas de SDCs, esto es, numerosas HKs y RRs diferentes que forman parte de sistemas de señalización aislados que detectan una señal específica y regulan en consecuencia determinados procesos celulares. Las diferentes HKs son proteínas homólogas entre sí, ya que catalizan el mismo tipo de reacción (fosforilación), y a su vez, las RRs son homólogas ya que son sustratos de las quinasas y, en su mayoría, son reguladores transcripcionales. Viendo este escenario, es difícil pensar que puede haber especificidad de manera que cada SDCs se mantenga aislado del resto. Por otra parte, si a un estímulo debe corresponder una respuesta, dicha especificidad debe ser estrictamente respetada. De hecho, una de las características más importantes de los SDCs bacterianos es la especificidad. Como se mencionó anteriormente, cada HK tiene una RR cognada, que constituye su pareja en un circuito de señalización. Y, en efecto, la fosforilación entre estas parejas de proteínas es rigorosamente específica in vivo, de manera que no existe una respuesta producida por un estímulo diferente. Diversos factores impiden que exista una fosforilación cruzada (conocida como cross-talk) entre una HK y una RR que no forman parte del mismo SDC: i) en primer lugar hay una especificidad, dada por la secuencia y la estructura tridimensional, en la interacción proteína-proteína necesaria para la transferencia de un grupo ~P [54, 55], ii) dicha especificidad redunda en una preferencia cinética en la interacción entre HK y RR cognados, en comparación con la de HK y RR no cognados, en otras palabras, hay competencia entre HKs por interactuar con una RR, y esta competencia la “gana” la HK que tenga mayor afinidad [15, 54–56], y iii) la actividad fosfatasa específica de la mayoría de las HKs en su estado inactivo suprime rápidamente cualquier efecto producido por la fosforilación de la RR cognada por una vía diferente, que puede ser proveniente de otra HK o de alguna molécula capaz de fosforilar RRs de manera no enzimática como el acetil-fosfato (acetil-P) [23, 56–59] .
“Inteligencia” bacteriana
Los mecanismos mencionados aseguran que haya una respuesta específica a un estímulo determinado, suprimiendo la relevancia fisiológica de reacciones de fosforilación cruzada entre proteínas no cognadas. Sin embargo, esto no quiere decir que todos los SDCs sean circuitos lineares. En efecto, existen sistemas en donde más de una HK fosforila específicamente a una sola RR, o por el contrario, una sola HK fosforila a varias RRs. A este tipo de sistemas se los conoce como ramificados (Fig. 3), y a pesar de tener esta característica, las interacciones y fosforilaciones entre sus componentes están finamente controladas para producir una respuesta determinada ante la presencia de un estímulo [55]. Existen muchos ejemplos de este tipo de sistemas en bacterias, algunos de los cuales son el sistema NblS/RpaB-SrrA de la cianobacteria Synechococcus elongates (una HK, dos RRs) [60], y el sistema de percepción de quórum de Vibrio harveyi, en donde las HKs LuxN, LuxQ y CqsS fosforilan a un único RR LuxO en respuesta a diferentes autoinductores [61] (Fig. 3).
Además, dos SDCs diferentes pueden estar conectados entre sí sin que haya reacciones de fosforilación cruzada. A este tipo de interacciones se las conoce como regulación cruzada, y están frecuentemente intermediadas por proteínas auxiliares que funcionan como conectores [62, 63]. El funcionamiento de estos sistemas se puede resumir de la siguiente manera: la expresión de un conector está regulada por un SDC (SDC1) y este conector se une a otra HK (HK2) o a una RR (RR2) que forman parte de otro SDC (SDC2), afectando su actividad quinasa o la estabilidad de la RR2-P. De esta manera, la presencia de una señal específica para el SDC1 afecta la respuesta del SDC2. Sin embargo, si no está presente la señal específica del SDC2, no hay una respuesta específica para este sistema. Así, las proteínas conectoras, en vez de contribuir a la inespecificidad de los SDCs, ayudan a la modulación de la respuesta coordinada por la presencia de varios estímulos diferentes. Ejemplos de estos sistemas interconectados son los SDCs PhoQ/PhoP y PmrB/PmrA de Salmonella enterica [64], conectados por la proteína PmrD; los SDCs CpxA/CpxR y EnvZ/OmpR de E. coli conectados por la proteína MzrA [65]; y PhoQ/PhoP y EvgA/EvgS de E. coli, que están conectados por SafA [66].
Este escenario, en donde existen SDCs ramificados y en donde muchos sistemas de transducción de señales en una célula bacteriana están interconectados funcionalmente entre sí, bajo un delicado control que asegura respuestas adecuadas y moduladas ante los cambios en las condiciones externas, trae a la memoria a las redes neuronales, propias de organismos superiores [67, 68]. Estas redes fosfo-neuronales bacterianas proporcionan a las bacterias, no sólo como organismos unicelulares sino también como comunidades bacterianas, comportamientos que podríamos atribuir como propios de una inteligencia bacteriana.
SDCs en organismos eucariotas
Como se mencionó en líneas anteriores, podemos encontrar SDCs en organismos tan diversos como arqueobacterias, levaduras, hongos filamentosos y plantas. El estudio de la evolución de estos sistemas en diferentes organismos constituye un tópico de gran interés, y actualmente se ve facilitado por la amplia colección de datos genómicos que permiten realizar estudios filogenéticos para determinar, por ejemplo, el origen de los diferentes módulos de un SDCs. Además, estos datos posibilitan indagar sobre la manera en que estos sistemas, con alta prevalencia en bacterias, pudieron ser adquiridos y adoptados por células eucariotas e incluso organismos superiores. A pesar de que la descripción de los aspectos evolutivos que circundan a los SDCs no es el objetivo de esta revisión, no podemos dejar de mencionar que la presencia de sistemas de transducción de señales basados en la fosforilación y trans-fosforilación de residuos de His y Asp en células eucariotas (y también en Arqueas) obedece a múltiples e independientes eventos de transferencia horizontal desde las bacterias [5, 13, 33, 69]. Sin embargo, cada organismo adoptó estos sistemas modulares de transducción de señales de una manera diferente, lo que se ve reflejado en diferencias importantes en la arquitectura y funcionamiento de los SDCs en organismos de diferentes reinos [15].
En plantas, es posible encontrar múltiples proteínas homólogas a las de los SDCs bacterianos, como histidín-quinasas (tanto HK como HHK), proteínas HPt y RRs. Sin embargo la distribución de estas proteínas es notablemente diferente a la de las bacterianas, ya que una célula vegetal suele tener muchas más RRs que HK/HHK (Fig. 3). Además, todas las HHK son bipartitas, conteniendo un DT y un DR [70]. En particular, Arabidopsis thaliana contiene 16 HK/HHK, 6 HPts y 32 RRs, aunque muchas de estas proteínas no son funcionales como parte de un SDC, ya que carecen de alguno de los residuos de Asp o His, indispensables para la fosforilación típica de estos sistemas. Así, sólo le quedan 8 HK/HHK, 5 HPts y 23 RRs con la capacidad de llevar a cabo un fosforelevo [70]. En realidad, hoy se sabe que 6 de estas 8 histidin-quinasas, todas ellas híbridas, y la totalidad de las proteínas HPTs y RRs de A. thaliana están involucradas en la detección de un grupo de hormonas vegetales conocidas como citoquininas, en donde la detección de estas señales desencadena la autofosforilación de al menos 3 HHK y un fosforelevo en donde participan las 5 HPts y los 23 RRs [71], de una manera que podría parecer redundante ya que múltiples proteínas llevan a cabo la misma tarea en la detección de una señal y para producir una misma respuesta, a diferencia de los SDCs bacterianos. Interesantemente, las otras 10 HK/HHK de A. thaliana tienen funciones de detección de estímulos y señalización pero con un mecanismo de acción diferente al de los SDCs, involucrando la asociación con serin-treonin-quinasas (receptores de etileno) [72] o la interacción directa con factores transcripcionales en el núcleo (fitocromos, que son HK no anclados a membranas) [73]. Interesantemente, proteínas típicas de SDCs bacterianos han sido integradas a sistemas de transducción de señales propios de eucariotas de tal manera que algunas de estas perdieron su actividad original encontrando nuevas funciones en los organismos que las adquirieron.
Otro ejemplo de esta adaptación de SDCs lo constituyen los hongos filamentosos, que se caracterizan por tener múltiples histidín-quinasas (hasta 21 en el caso de Cochliobolus heterostrophus) [11], todas ellas híbridas y bipartitas, y una única proteina HPt, capaz de atravesar la membrana nuclear [74] (Fig. 3). Además la gran mayoría de las HHK de hongos son proteínas solubles, contrario a lo que ocurre en bacterias, lo que deja abierta la posibilidad de que estas también puedan entrar al núcleo. Finalmente, en estas células suelen existir dos reguladores de respuesta, uno de los cuales tiene un dominio de unión a DNA y el otro interactúa con proteínas involucradas en la vía de las MAP quinasas (MAPK), ruta de señalización eucariota basada en fosforilaciones en residuos de treonina y tirosina [75]. La arquitectura de este SDC es convergente, ya que múltiples quinasas perciben igual número de estímulos, y todas ellas fosforilan a un único HPt, que a su vez es capaz de fosforilar a dos RRs (Fig. 3). Esta distribución no permite visualizar fácilmente cómo podría haber una especificidad que asegure una respuesta diferente ante cada estímulo. Aunque este interrogante no ha sido aún resuelto, se puede especular sobre posibles mecanismos que aseguren la especificidad, necesaria para cualquier sistema de transducción de señales. Es posible que existan proteínas accesorias que promuevan la interacción de algunos componentes bajo determinadas condiciones, por ejemplo una HHK con el único HPt y uno de los dos RRs, aislando a estas proteínas de otras interacciones. Sin embargo, aun con proteínas accesorias, solo podrían visualizarse dos posibles respuestas dadas por los dos RRs. Otra posibilidad es que algunas HHKs transduzcan la señal mediante un mecanismo independiente de fosforilación del HPt o de las RRs, como ocurre en el caso de los receptores de etileno de plantas. Finamente, hay algunas evidencias que sugieren que algunas HHK, por su naturaleza soluble, pueden ingresar al núcleo e interactuar con proteínas reguladoras de la expresión génica como factores transcripcionales o proteínas modificadoras de histonas [76, 77]. La conjunción de todos estos factores podría resultar en sistemas de señalización específicos, como los SDCs bacterianos, pero con modos de acción sustancialmente diferentes, como ocurre en plantas, en donde no siempre participa la transferencia del ~P entre His y Asp.
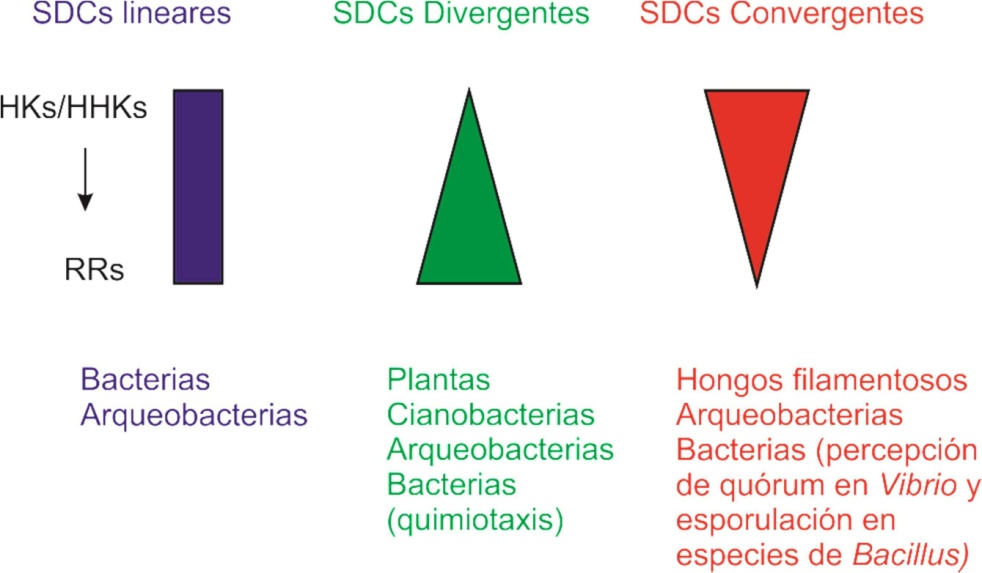
Figura 3: Arquitecturas encontradas en SDCs de diferentes organismos. La mayoría de los SDCs bacterianos son lineares (una HK/HHK con una RR); los SDCs de cianobacterias y plantas se caracterizan por tener más RRs que HKs/HHKs, son sistemas ramificados divergentes; mientras que los SDCs de hongos filamentosos son altamente convergentes, con muchas HHKs y pocas RRs. Algunos sistemas bacterianos presentan arquitecturas convergentes o divergentes, como son el sistema quimiotáctico bacteriano (divergente) y los sistemas de percepción de quórum (especies de Vibrio) y de señalización de la esporulación en especies de Bacillus (convergentes). En arqueobacterias pueden encontrarse las tres arquitecturas de SDCs.
Consideraciones finales
Los SDCs son la principal vía de comunicación de las células bacterianas con el entorno, y les permite adaptarse rápidamente a cualquier cambio en las condiciones existentes. Estos sistemas, basados en transferencias de ~P entre residuos de His y Asp, han sido tan exitosos que han proliferado y prevalecido en el reino de las bacterias y han sido, además, adquiridos por arqueobacterias y células eucariotas, aunque en estas últimas adoptaron funciones y características distintivas que se adecúan a las necesidades propias de cada tipo de organismo.
Quizás la propiedad más importante de los SDCs bacterianos es la especificidad, la cual es rigurosamente conservada mediante múltiples mecanismos, que impiden una fosforilación cruzada entre diferentes SDCs. Sin embargo, existe comunicación controlada entre distintos sistemas contenidos en una misma célula, de manera que se conforman redes de señalización que permiten a las bacterias regular finamente la respuesta ante oleadas de estímulos a los que generalmente están sujetas.
Referencias:
1. Nixon BT, Ronson CW, Ausubel FM (1986) Two-component regulatory systems responsive to environmental stimuli share strongly conserved domains with the nitrogen assimilation regulatory genes ntrB and ntrC. Proc. Natl. Acad. Sci. U. S. A. 83: 7850–7854.
2. Ronson CW, Nixon BT , Ausubel FM (1987) Conserved domains in bacterial regulatory proteins that respond to environmental stimuli. Cell 49: 579–581.
3. Galperin MY (2005) A census of membrane-bound and intracellular signal transduction proteins in bacteria: bacterial IQ, extroverts and introverts. BMC Microbiol. 5: 35 DOI: 10.1186/1471-2180-5-35.
4. Ulrich LE, Zhulin IB (2010) The MiST2 database: a comprehensive genomics resource on microbial signal transduction. Nucleic Acids Res. 38: D401-407 DOI: 10.1093/nar/gkp940.
5. Wuichet K, Cantwell BJ, Zhulin IB (2010) Evolution and phyletic distribution of two- component signal transduction systems. Curr. Opin. Microbiol. 13: 219–225 DOI: 10.1016/j.mib.2009.12.011.
6. Stover CK, Pham XQ, Erwin AL, Mizoguchi SD, Warrener P, Hickey MJ et al. (2000) Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen. Nature 406: 959–964 DOI: 10.1038/35023079.
7. Blattner FR, Plunkett G, a Bloch C, Perna NT, Burland V, Riley M, et al. (1997) The complete genome sequence of Escherichia coli K-12. Science 277: 1453–1462.
8. Ashby MK (2006) Distribution, structure and diversity of “bacterial” genes encoding two- component proteins in the Euryarchaeota. Archaea 2: 11–30 DOI: 10.1155/2006/562404.
9. Thomason P, Kay R (2000) Eukaryotic signal transduction via histidine-aspartate phosphorelay. J. Cell Sci. 113: 3141–3150.
10. Santos JL, Shiozaki K (2001) Fungal histidine kinases. Sci. STKE 2001: re1 DOI: 10.1126/stke.2001.98.re1.
11. Catlett NL, Yoder OC, Turgeon BG (2003) Whole-genome analysis of two- component signal transduction genes in fungal pathogens. Eukaryot. Cell 2: 1151–1161.
12. Schaller GE, Shiu SH, Armitage JP (2011) Two-component systems and their co- option for eukaryotic signal transduction. Curr. Biol. 21: R320-330 DOI: 10.1016/j.cub.2011.02.045.
13. Koretke KK, Lupas AN, Warren PV, Rosenberg M, Brown JR (2000) Evolution of two-component signal transduction. Mol. Biol. Evol. 17: 1956–1970.
14. Wolanin PM, Thomason PA, Stock JB (2002) Histidine protein kinases: key signal transducers outside the animal kingdom. Genome Biol. 3: REVIEWS3013.
15. Alvarez AF, Barba-Ostria C, Silva-Jiménez H, Georgellis D (2016) Organization and mode of action of two component system signaling circuits from the various kingdoms of life. Environ. Microbiol. 18: 3210–3226 DOI: 10.1111/1462-2920.13397.
16. Kofoid EC, and JS Parkinson (1988) Transmitter and receiver modules in bacterial signaling proteins. Proc. Natl. Acad. Sci. U. S. A. 85: 4981–4985.
17. Dutta R, Qin L, Inouye M (1999) Histidine kinases: diversity of domain organization. Mol. Microbiol. 34: 633–640.
18. Levit M, Liu Y, Surette M, Stock J (1996) Active Site Interference and Asymmetric Activation in the Chemotaxis Protein Histidine Kinase CheA. J. Biol. Chem. 271: 32057–32063 DOI: 10.1074/jbc.271.50.32057.
19. Tanaka T, Saha SK, Tomomori C, Ishima R, Liu D, Tong KI, et al. (1998) NMR structure of the histidine kinase domain of the E. coli osmosensor EnvZ. Nature 396: 88–92 DOI: 10.1038/23968.
20. West AH, Stock AM (2001) Histidine kinases and response regulator proteins in two- component signaling systems. Trends Biochem. Sci. 26: 369–376.
21. Cheung J, Hendrickson WA (2010) Sensor domains of two-component regulatory systems. Curr. Opin. Microbiol. 13: 116–123 DOI: 10.1016/j.mib.2010.01.016.
22. Stock AM, Robinson VL, Goudreau PN (2000) Two-component signal transduction. Annu. Rev. Biochem. 69: 183–215 DOI: 10.1146/annurev.biochem.69.1.183.
23. Kenney LJ (2010) How important is the phosphatase activity of sensor kinases? Curr. Opin. Microbiol. 13: 168–176 DOI: 10.1016/j.mib.2010.01.013.
24. Burbulys D, Trach KA, Hoch JA (1991) Initiation of sporulation in B. subtilis is controlled by a multicomponent phosphorelay. Cell 64: 545–552.
25. Uhl MA, Miller JF (1996) Integration of multiple domains in a two-component sensor protein: the Bordetella pertussis BvgAS phosphorelay. EMBO J. 15: 1028–1036.
26. Georgellis D, Lynch AS, Lin EC (1997) In vitro phosphorylation study of the Arc two-component signal transduction system of Escherichia coli. J. Bacteriol. 179: 5429–5435.
27. Takeda S, Fujisawa Y, Matsubara M, Aiba H, Mizuno T (2001) A novel feature of the multistep phosphorelay in Escherichia coli: a revised model of the RcsC --> YojN --> RcsB signalling pathway implicated in capsular synthesis and swarming behaviour. Mol. Microbiol. 40: 440–450.
28. Williams RHN, Whitworth DE (2010) The genetic organisation of prokaryotic two- component system signalling pathways. BMC Genomics 11: 720 DOI: 10.1186/1471-2164-11-720.
29. Uhl MA, Miller JF (1996) Central role of the BvgS receiver as a phosphorylated intermediate in a complex two-component phosphorelay. J. Biol. Chem. 271: 33176–33180.
30. Georgellis D, Kwon O, De Wulf P, Lin EC (1998) Signal decay through a reverse phosphorelay in the Arc two-component signal transduction system. J. Biol. Chem. 273: 32864– 32869.
31. Peña-Sandoval GR, Kwon O, Georgellis D (2005) Requirement of the receiver and phosphotransfer domains of ArcB for efficient dephosphorylation of phosphorylated ArcA in vivo. J. Bacteriol. 187: 3267–3272.
32. Kwon O, Georgellis D, Lin EC (2000) Phosphorelay as the sole physiological route of signal transmission by the Arc two-component system of Escherichia coli. J. Bacteriol. 182: 3858–3862.
33. Capra EJ, Laub MT (2012) Evolution of two-component signal transduction systems. Annu. Rev. Microbiol. 66: 325–347 DOI: 10.1146/annurev-micro-092611-150039.
34. Krell T, Lacal J, Busch A, Silva-Jiménez H, Guazzaroni ME, Ramos JL (2010) Bacterial sensor kinases: diversity in the recognition of environmental signals. Annu. Rev. Microbiol. 64: 539–559 DOI: 10.1146/annurev.micro.112408.134054.
35. Möglich A, Ayers RA, Moffat K (2009) Structure and Signaling Mechanism of Per- ARNT-Sim Domains. Structure 17: 1282–1294 DOI: 10.1016/j.str.2009.08.011.
36. Taylor BL, Zhulin IB (1999) PAS domains: internal sensors of oxygen, redox potential, and light. Microbiol. Mol. Biol. Rev. 63: 479–506.
37. Zhulin IB, Taylor BL, Dixon R (1997) PAS domain S-boxes in Archaea, Bacteria and sensors for oxygen and redox. Trends Biochem. Sci. 22: 331–333.
38. Kaspar S, Perozzo R, Reinelt S, Meyer M, Pfister K, Scapozza L, et al. (1999) The periplasmic domain of the histidine autokinase CitA functions as a highly specific citrate receptor. Mol. Microbiol. 33: 858–872.
39. Pappalardo L, Janausch IG, Vijayan V, Zientz E, Junker J, Peti W, et al. (2003) The NMR structure of the sensory domain of the membranous two-component fumarate sensor (histidine protein kinase) DcuS of Escherichia coli. J. Biol. Chem. 278: 39185–39188 DOI: 10.1074/jbc.C300344200.
40. Reinelt S, Hofmann E, Gerharz T, Bott M, Madden DR (2003) The structure of the periplasmic ligand-binding domain of the sensor kinase CitA reveals the first extracellular PAS domain. J. Biol. Chem. 278: 39189–39196 DOI: 10.1074/jbc.M305864200.
41. Kneuper H, Janausch IG, Vijayan V, Zweckstetter M, Bock V, Griesinger C, et al. (2005) The nature of the stimulus and of the fumarate binding site of the fumarate sensor DcuS of Escherichia coli. J. Biol. Chem. 280: 20596–20603 DOI: 10.1074/jbc.M502015200.
42. Cho US, Bader MW, Amaya MF, Daley ME, Klevit RE, Miller SI, et al. (2006) Metal bridges between the PhoQ sensor domain and the membrane regulate transmembrane signaling. J. Mol. Biol. 356: 1193–1206 DOI: 10.1016/j.jmb.2005.12.032.
43. Miyatake H, Mukai M, Park SY, Adachi S, Tamura K, Nakamura H, et al. (2000) Sensory mechanism of oxygen sensor FixL from Rhizobium meliloti: crystallographic, mutagenesis and resonance raman spectroscopic studies. J. Mol. Biol. 301: 415–431 DOI: 10.1006/jmbi.2000.3954.
44. Liu X, De Wulf P (2004) Probing the ArcA-P modulon of Escherichia coli by whole genome transcriptional analysis and sequence recognition profiling. J. Biol. Chem. 279: 12588– 12597.
45. Salmon KA, Hung S, Steffen NR, Krupp R, Baldi P, Hatfield GW, et al. (2005) Global gene expression profiling in Escherichia coli K12: effects of oxygen availability and ArcA. J. Biol. Chem. 280: 15084–15096 DOI: 10.1074/jbc.M414030200.
46. Matsushika A, Mizuno T (2000) Characterization of three putative sub-domains in the signal-input domain of the ArcB hybrid sensor in Escherichia coli. J. Biochem. 127: 855–860.
47. Malpica R, Sandoval GR, Rodriguez C, Franco B, Georgellis D (2006) Signaling by the Arc two-component system provides a link between the redox state of the quinone pool and gene expression. Antioxid. Redox Signal 8: 781–795.
48. Malpica R, Franco B, Rodriguez C, Kwon O, Georgellis D (2004) Identification of a quinone-sensitive redox switch in the ArcB sensor kinase. Proc. Natl. Acad. Sci. U S A 101: 13318– 13323.
49. Georgellis D, Kwon O, Lin EC (2001) Quinones as the redox signal for the Arc two- component system of bacteria. Science (80-. ). 292: 2314–2316.
50. Alvarez AF, Rodriguez C, Georgellis D (2013) Ubiquinone and menaquinone electron carriers represent the yin and yang in the redox regulation of the ArcB sensor kinase. J. Bacteriol. 195: 3054–3061 DOI: 10.1128/JB.00406-13.
51. Ulrich LE, Koonin EV, Zhulin IB (2005) One-component systems dominate signal transduction in prokaryotes. Trends Microbiol. 13: 52–56 DOI: 10.1016/j.tim.2004.12.006.
52. Aguilar PS, Hernandez-Arriaga AM, Cybulski LE, Erazo AC, de Mendoza D (2001) Molecular basis of thermosensing: a two-component signal transduction thermometer in Bacillus subtilis. EMBO J. 20: 168116–91 DOI: 10.1093/emboj/20.7.1681.
53. Galperin MY (2010) Diversity of structure and function of response regulator output domains. Curr. Opin. Microbiol. 13: 150–159 DOI: 10.1016/j.mib.2010.01.005.
54. Skerker JM, Perchuk BS, Siryaporn A, Lubin EA, Ashenberg O, Goulian M, et al. (2008) Rewiring the specificity of two-component signal transduction systems. Cell 133: 1043–1054 DOI: 10.1016/j.cell.2008.04.040.
55. Laub MT, Goulian M (2007) Specificity in two-component signal transduction pathways. Annu. Rev. Genet. 41: 121–145 DOI: 10.1146/annurev.genet.41.042007.170548.
56. Groban ES, Clarke EJ, Salis HM, Miller SM, Voigt CA (2009) Kinetic buffering of cross talk between bacterial two-component sensors. J. Mol. Biol. 390: 380–393 DOI: 10.1016/j.jmb.2009.05.007.
57. Guckes KR, Kostakioti M, Breland EJ, Gu AP, Shaffer CL, Martinez CR, et al. (2013) Strong cross-system interactions drive the activation of the QseB response regulator in the absence of its cognate sensor. Proc. Natl. Acad. Sci. U. S. A. 110: 16592–16597 DOI: 10.1073/pnas.1315320110.
58. Siryaporn A, Goulian M (2008) Cross-talk suppression between the CpxA-CpxR and EnvZ-OmpR two-component systems in E. coli. Mol. Microbiol. 70: 494–506 DOI: 10.1111/j.1365- 2958.2008.06426.x.
59. Huynh TN, Stewart V (2011) Negative control in two-component signal transduction by transmitter phosphatase activity. Mol. Microbiol. 82: 275–286 DOI: 10.1111/j.1365- 2958.2011.07829.x.
60. López-Redondo ML, Moronta F, Salinas P, Espinosa J, Cantos R, Dixon R, et al. (2010) Environmental control of phosphorylation pathways in a branched two-component system. Mol. Microbiol. 78: 475–489.
61. Henke JM, Bassler BL (2004) Three parallel quorum-sensing systems regulate gene expression in Vibrio harveyi. J. Bacteriol. 186: 6902–6914 DOI: 10.1128/JB.186.20.6902- 6914.2004.
62. Wanner BL (1992) Is cross regulation by phosphorylation of two-component response regulator proteins important in bacteria? J. Bacteriol. 174: 2053–2058.
63. Eguchi Y, Utsumi R (2005) A novel mechanism for connecting bacterial two- component signal-transduction systems. Trends Biochem. Sci. 30: 70–72 DOI: 10.1016/j.tibs.2004.12.003.
64. Kato A, Groisman EA (2004) Connecting two-component regulatory systems by a protein that protects a response regulator from dephosphorylation by its cognate sensor. Genes Dev. 18: 2302–2313 DOI: 10.1101/gad.1230804.
65. Gerken H, Charlson ES, Cicirelli EM, Kenney LJ, Misra R (2009) MzrA: a novel modulator of the EnvZ/OmpR two-component regulon. Mol. Microbiol. 72: 1408–1422 DOI: 10.1111/j.1365-2958.2009.06728.x.
66. Eguchi Y, Ishii E, Hata K, Utsumi R (2010) Regulation of Acid Resistance by Connectors of Two-Component Signal Transduction Systems in Escherichia coli. J. Bacteriol. 193: 1222–1228 DOI: 10.1128/JB.01124-10.
67. Hellingwerf KJ, Postma PW, Tommassen J, Westerhoff HV (1995) Signal transduction in bacteria: phospho-neural network(s) in Escherichia coli? FEMS Microbiol. Rev. 16.
68. Armitage JP, Holland IB, Jenal U, Kenny B (2005) “Neural Networks” in Bacteria: Making Connections. J. Bacteriol. 187: 26–36 DOI: 10.1128/JB.187.1.26-36.2005.
69. Kim D, Forst S (2001) Genomic analysis of the histidine kinase family in bacteria and archaea. Microbiology 147: 1197–1212 DOI: 10.1099/00221287-147-5-1197.
70. Hwang I, Chen HC, Sheen J (2002) Two-component signal transduction pathways in Arabidopsis. Plant Physiol. 129: 500–515 DOI: 10.1104/pp.005504.
71. Kakimoto T (2003) Perception and signal transduction of cytokinins. Annu. Rev. Plant Biol. 54: 605–627 DOI: 10.1146/annurev.arplant.54.031902.134802.
72. Shakeel SN, Wang X, Binder BM, Schaller GE (2013) Mechanisms of signal transduction by ethylene: overlapping and non-overlapping signalling roles in a receptor family. AoB Plants 5: plt010 DOI: 10.1093/aobpla/plt010.
73. Ni M, Tepperman JM, Quail PH (1998) PIF3, a phytochrome-interacting factor necessary for normal photoinduced signal transduction, is a novel basic helix-loop-helix protein. Cell 95: 657–667.
74. Lu JMY, Deschenes RJ, Fassler JS (2003) Saccharomyces cerevisiae histidine phosphotransferase Ypd1p shuttles between the nucleus and cytoplasm for SLN1-dependent phosphorylation of Ssk1p and Skn7p. Eukaryot. Cell 2: 1304–1314.
75. Posas F, Wurgler-Murphy SM, Maeda T, Witten EA, Thai TC, Saito H (1996) Yeast HOG1 MAP kinase cascade is regulated by a multistep phosphorelay mechanism in the SLN1-YPD1-SSK1 “two-component” osmosensor. Cell 86: 865–875.
76. Foureau E, Clastre M, Obando Montoya EJ, Besseau S, Oudin A, Glévarec G, et al. (2014) Subcellular localization of the histidine kinase receptors Sln1p, Nik1p and Chk1p in the yeast CTG clade species Candida guilliermondii. Fungal Genet. Biol. 65: 25–36 DOI: 10.1016/j.fgb.2014.01.007.
77. Hedtke M, Rauscher S, Röhrig J, Rodríguez-Romero J, Yu Z, Fischer R (2015) Light-dependent gene activation in Aspergillus nidulans is strictly dependent on phytochrome and involves the interplay of phytochrome and white collar-regulated histone H3 acetylation. Mol. Microbiol. 97: 733–745 DOI: 10.1111/mmi.13062.
Adrián F. Álvarez es investigador asociado y Dimitris Georgellis es investigador principal, ambos de la Universidad Nacional Autónoma de México
|
Revista QuímicaViva Número 3, año 15, Diciembre Agosto 2016 quimicaviva@qb.fcen.uba.ar |