RNA virus, emergencia y coronavirus
Sandra M. Cordo
Departamento de Química Biológica, Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires, IQUIBICEN, CONICET, Buenos Aires, Argentina.
Generalidades de los RNA virus
La naturaleza del genoma de un virus y la estrategia para la síntesis del mRNA es la base de la “Clasificación Baltimore”. Esta clasificación es práctica y divide a los virus en 7 grupos según posean genoma a DNA o RNA, el sentido (o polaridad) de sus moléculas sea positivo o negativo y estas se presenten en simple o doble cadena. La clasificación no es taxonómica, aunque las categorías taxonómicas reconocidas por ICTV (International Committee on Taxonomy of Viruses), por ejemplo Familias, pueden acomodarse dentro de estos 7 grupos.
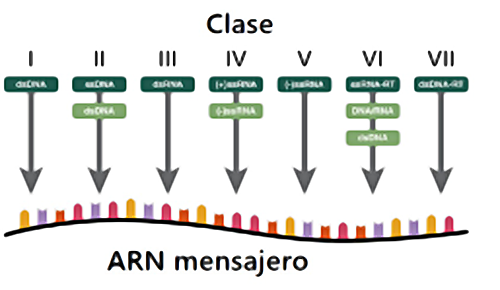
Figura #: Clasificación de Baltimore según el genoma viral. Existen siete clases de genomas virales que pueden ser de DNA o RNA, y al mismo tiempo monocatenarios (ss) o bicatenarios (ds). Se muestra la ruta que toma cada tipo de virus para formar su mRNA (ARN mensajero) y la estrategia para su replicación (adaptado de Madigan et al., 2015)
Los virus animales a RNA, entonces, comprenden los Grupos III, IV y V de esta clasificación (Figura 1). El Grupo III contiene los virus de RNA doble cadena a partir del cual son sintetizados los mRNA. En el Grupo IV se encuentran aquellos virus con RNA de sentido positivo capaz de funcionar directamente como mRNA. El Grupo V contiene los virus con RNA de sentido negativo a partir del cual deben sintetizar sus mRNAs. Algunas de las Familias virales de importancia sanitaria humana en cada Grupo pueden observarse en la Tabla 1.
| Grupo | Familia | Virus | Reservorio | Enfermedad |
|---|---|---|---|---|
| III | Reoviridae | Rotavirus | Humanos | Gastroenteritis, diarrea |
| IV | Flaviviridae | Dengue virus | Monos, humanos | Fiebre hemorrágica |
| West Nile virus | Aves | Encefalitis | ||
| Zika virus | Monos, ovejas, patos, caballos,murciélagos | Fiebre de Zika, microcefalia congénita | ||
| Coronaviridae | HCoV (Coronavirus-humano) | Murciélagos, roedores | Enfermedad respiratoria leve | |
| SARS-CoV (síndrome agudo respiratorio-coronavirus) | Murciélagos | Síndrome respiratorio agudo | ||
| MERS-CoV (síndrome respiratorio del Medio Este-coronavirus) | Murciélagos | Síndrome respiratorio agudo | ||
| V | Bunyaviridae | Hantaan virus | Ratones salvajes | Fiebre hemorrágica |
| Sin Nombre virus | Ratones salvajes | Síndrome pulmonar | ||
| Orthomyxoviridae | Influenza H5N1 | Aves salvajes | Enfermedad respiratoria (Vietman, 2003) | |
| Influenza H7N9 | Aves salvajes | Enfermedad respiratoria (China, 2013) | ||
| Paramyxoviridae | Hendra virus | Murciélagos | Hemorragia pulmonar | |
| Nipha virus | Murciélagos | Encefalitis | ||
| Rabdoviridae | Rabia virus | Murciélagos | Síndrome neurológico agudo | |
| Mammarenaviridae | Lassa virus | Ratones salvajes | Fiebre hemorrágica | |
| Junín virus | Ratones salvajes | Fiebre hemorrágica | ||
| Filoviridae | Ebola virus | Murciélagos | Fiebre hemorrágica |
Tabla I: Familias virales de importancia sanitaria humana con genoma RNA. Se muestran algunas de las familias de virus RNA más relevantes para la salud humana. Se mencionan el virus particular, su reservorio en la naturaleza y la enfermedad que produce en el humano.
En cualquier caso, un virus a RNA es un virus que utiliza la RNA polimerasa RNA dependiente (RdRp) para replicar o sintetizar su genoma. Esta polimerasa es siempre codificada por el genoma viral y en algunas Familias forma parte del virión, siendo una proteína estructural (Grupo V). Durante la replicación de los virus a RNA al menos tres tipos de RNA deben ser sintetizados: el genoma, la copia del genoma y los mensajeros. Algunas Familias de virus además sintetizan RNA subgenómicos, y en todos estos procesos se necesitará una RdRp.
Una característica saliente de los virus en general es su alta tasa de mutación. Durante el ciclo replicativo de un virus RNA la tasa es particularmente alta debido a la falta de actividad de corrección de la polimerasa viral en combinación con la naturaleza estructuralmente más flexible de las moléculas de RNA, en comparación con las de DNA. Por ejemplo, la tasa de mutación de un virus RNA se encuentra alrededor de 10-3 a 10-5 en comparación a los virus a DNA en el rango de 10-6 a 10-8 (Figura 2).
La implicancia de este fenómeno en el contexto del ambiente (viral) y sus fluctuaciones explica el proceso de “adaptación” o “evolución viral”. Los RNA virus pueden mutar y evolucionar rápidamente en contextos del estrés antiviral producidoporlacélula hospedadora, la respuesta inmune del organismo infectado y/o la exposición a tratamientos antivirales. De esta forma no sorprende que la mayoría de los nuevos virus emergentes pertenecen al grupo de RNA virus.
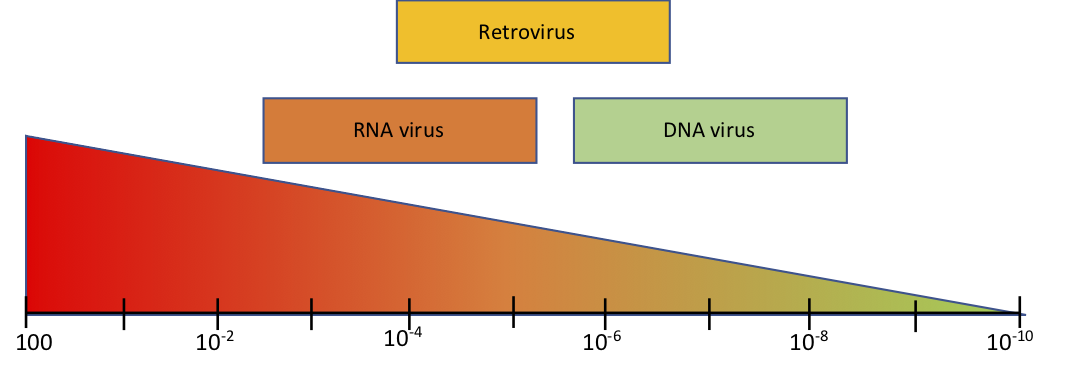
Figura 2: Tasa de mutación en genomas virales. Las tasas de mutación responden principalmente al tipo de molécula presente en el genoma viral. Los números se expresan como: mutación/sitio/ronda de replicación. La tasa de mutación de un virus RNA es de 1:1000 a 1:100000
Enfermedades y virus emergentes
Una enfermedad emergente es un término utilizado para describir la aparición de una infección previamente desconocida en una especie en particular, o una infección ya conocida pero que se presenta o expande en un nuevo huésped, nicho ecológico, o zona geográfica y a menudo estas características están acompañadas de un aumento importante en la patogenicidad. Ejemplos recientes de este fenómeno son: el cruce de influenza aviaria a humanos en el año 1995 (H5N1), la expansión del virus Ébola hacia África Occidental (2014) o la emergencia del virus Zika (originalmente conocido como una enfermedad febril en África Oriental) en zonas tan lejanas como Polinesia y Sudamérica. Más recientemente, por supuesto, la emergencia del coronavirus SARS en 2003 y 2019.
Familia Coronaviridae
Dentro del Grupo IV de la Clasificación Baltimore, la Familia Coronaviridae incluye 3 géneros con virus ampliamente distribuidos en huéspedes tan variados como el humano, mamíferos pequeños, aves y otros, causando infecciones tanto agudas como persistentes.
La mayoría de los coronavirus se transmiten a sus huéspedes susceptibles por la ruta respiratoria o fecal, siendo el sitio inicial de replicación las células epiteliales. Algunos coronavirus humanos (HCoV) y también porcinos replican principalmente en las células epiteliales respiratorias causando infecciones restringidas y sintomatologías localizadas. Otros coronavirus (como el bovino, felino, canino) infectan células epiteliales del tracto entérico, causando diarreas agudas y a veces fatales.Los coronavirus felinos en ocasiones pueden producir infecciones sistémicas causando una enfermedad generalizada y muerte. Los coronavirus porcinos que causan predominantemente infecciones entéricas localizadas son sin embargo también neurotrópicos. Los coronavirus SARS-CoV y MERS-CoV son causantes de las más severas de las infecciones conocidas por coronavirus humanos.SARS-CoV-2 parece ser menos virulento (3-4 % de mortalidad reportado por WorldHealth Organization) en comparación con el SARS-CoV (10 % de mortalidad) y el MERS-CoV (35 % de mortalidad), con la excepción de la infección de SARS-CoV-2 en los ancianos y en aquellos con otras enfermedades de base. Este virus Infecta las vías respiratorias altas y bajas causando injurias de moderadas a severas en pulmones. El virus también es detectado en otros órganos como riñón, hígado e intestino.
Figura 3: Ilustración de la familia de los coronavirus.
El coronavirus humano HCoV-229E (del género Alfacoronavirus), capaz de producir enfermedades leves, utilizan como receptor celular hAPN (por sus siglas en inglés Human-aminopeptidase N). Una característica distintiva de SARS-CoV (del género Betacoronavirus) es que infecta las células blanco por medio del receptor ACE2 (por sus siglas en inglés Angiotensin-Converting Enzyme 2). Esta molécula es una proteína ubicada en la membrana celular e involucrada en la regulación de la presión sanguínea y la función cardíaca. En humanos se expresa en células epiteliales de pulmón e intestino delgado los cuales son los blancos primarios de SARS-CoV. ACE2 también se expresa en otros órganos como el corazón y riñones. Estudios estructurales han demostrado que la mutación de solo 2 residuos aminoacídicos en la proteína de superficie del SARS-CoV permitió el salto de especie: de la civeta asiática al humano. Recientemente se ha demostrado que esta proteína también funciona como receptor para el nuevo SARS-CoV-2. Sin embargo, aún no hay una clara relación entre patogénesis y receptor. Un estudio publicado recientemente sugiere que variantes genéticas de la proteína ACE2 en las distintas etnias poblacionales podrían presentar distintos niveles de afinidad y reconocimiento con SARS-CoV-2; y así explicar la severidad de la enfermedad en los distintos continentes.
Los distintos coronavirus humanos (HCoV-OC43, HCoV-229E, HCoV-NL63 y HCoV-HKU1) causan hasta el 30% de infecciones leves del tracto superior respiratorio considerándose endémicos de poblaciones humanas, desde los años 60 cuando el primero de ellos fue identificado. La emergencia y re-emergencia de nuevos RNA virus y en particular dentro de esta familia (SARS-CoV, MERS-CoV) está siendo más frecuente de lo esperado. Las características altamente transmisibles y patógenas de estas nuevas emergencias ponen en evidencia la urgencia de comprender en mayor profundidad los factores que facilitan éste fenómeno y acelerar los estudios que permitan mitigar y prevenir las consecuencias de estas infecciones en la salud humana.
Los coronavirus son virus envueltos de genoma no segmentado, es decir que la información genética se encuentra en una sola molécula de RNA. Esta única molécula de 26 Kb a 30 Kb de longitud es la más grande entre todos los virus de RNA, incluyendo aquellos cuyo genoma es segmentado. Sin embargo, la estrategia replicativa dista de ser sencilla y muestra características distintivas dentro del grupo. Todos sus miembros presentan una idéntica organización genómica que consta del gen de la replicasa rio arriba de los genes de las proteínas estructurales. El gen de la replicasa corresponde a dos tercios del total de la molécula y es el único que se traduce directamente del genoma, dando dos productos proteicos diferentes. Por un intrincado mecanismo de desplazamiento de los ribosomas celulares se traducen dos polipéptidos definidos pp1a (440–500 kDa) y pp1ab (740– 810 kDa). A partir de estos y por un proceso autoproteolítico se expresan las proteínas no estructurales (designadas nsp1 a nsp16) necesarias para formar el complejo replicasa-transcriptasa (RTC) y completar un ciclo de infección exitoso. Todos los eventos posteriores en la replicación de los coronavirus están determinados por la expresión inicial de la replicasa.
Referencias:
Brierley I, Digard P, Inglis SC (1989) Characterization of an eficient coronavirus ribosomal frameshifting signal: requirement for an RNA pseudo- knot. Cell 57:537–547.
Cao Yanan, Lin Li, Zhimin Feng, Shengqing Wan, Peide Huang, Xiaohui Sun, Fang Wen, Xuanlin Huang, Guang Ning &Weiqing Wang (2020) Comparative genetic analysis of the novel coronavirus (2019-nCoV/SARS-CoV-2) receptor ACE2 in different populations. Cell Discovery 6: 11,
Siobain D. (2018) “Why are RNA virus mutation rates so damn high?.” PLoS biology. doi:10.1371/journal.pbio.3000003
Knipe DM, Howley PM, editors (2013) Fields Virology (6th Edition). Philadelphia, USA: Lippincott Williams & Wilkins.
Guo YR, Cao QD, Hong ZS, Tan YY, Chen SD, Jin HJ, Tan KS, Wang DY, Yan Y (2020) The origin, transmission and clinical therapies on coronavirus disease 2019 (COVID-19) outbreak - an update on the status. Military Medical Research. 13;7(1):11. doi: 10.1186/s40779-020-00240-0.
Li F (2008) Structural analysis of major species barriers between humans and palm civets for severe acute respiratory syndrome coronavirus infections. Journal of Virology 82:6984–6991.
Li W, Zhang C, Sui J (2005) Receptor and viral determinants of SARS- coronavirus adaptation to human ACE2. EMBO Journal 24:1634–1643.
Li W, Moore MJ, Vasilieva N (2003) Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus. Nature 426: 450–454.
Masters PS (2006) The molecular biology of coronaviruses. Advances in Virus Research 66:193–292.
Wang-Shick, R (2017) Molecular Virology of Human Pathogenic Viruses. 1st. Edition. Academic Press, Elsevier. doi:10.1016/B978-0-12-800838-6.00016-3.
Cann, AJ (2016) Principles of Molecular Virology Sixth Edition. Academic Press, Elsevier.
Zhang H, Penninger JM, Li Y, Zhong N, Slutsky AS (2020) Angiotensin-converting enzyme 2 (ACE2) as a SARS-CoV-2 receptor: molecular mechanisms and potential therapeutic target. Intensive Care Medicine. 56:586-590 doi: 10.1007/s00134-020-05985-9.
|
Revista QuímicaViva Número 3, año 19, Abril 2020 quimicaviva@qb.fcen.uba.ar |